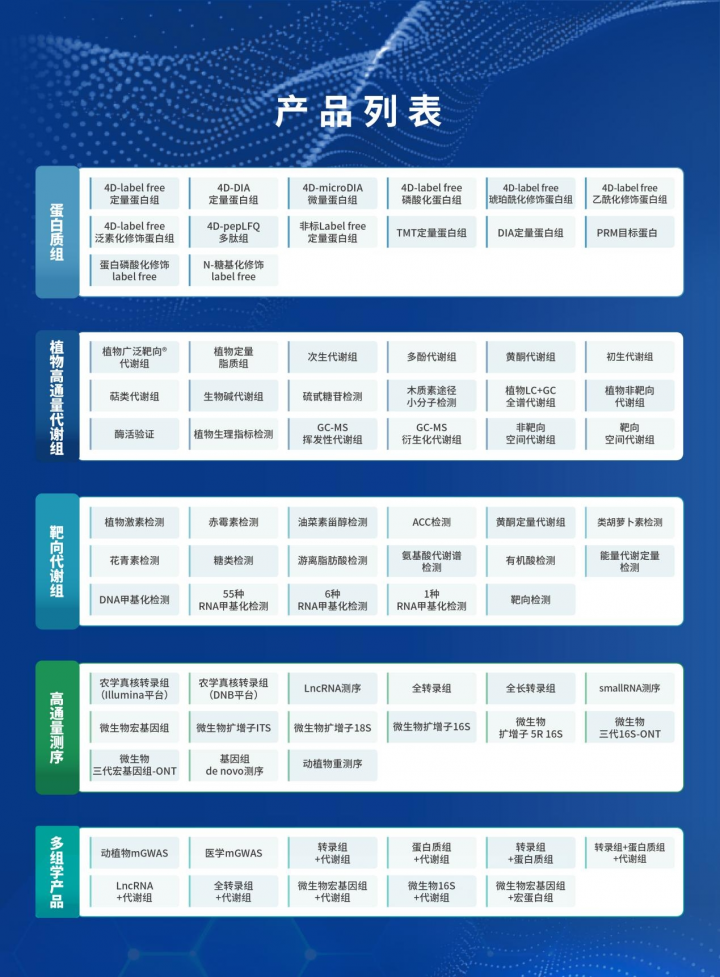
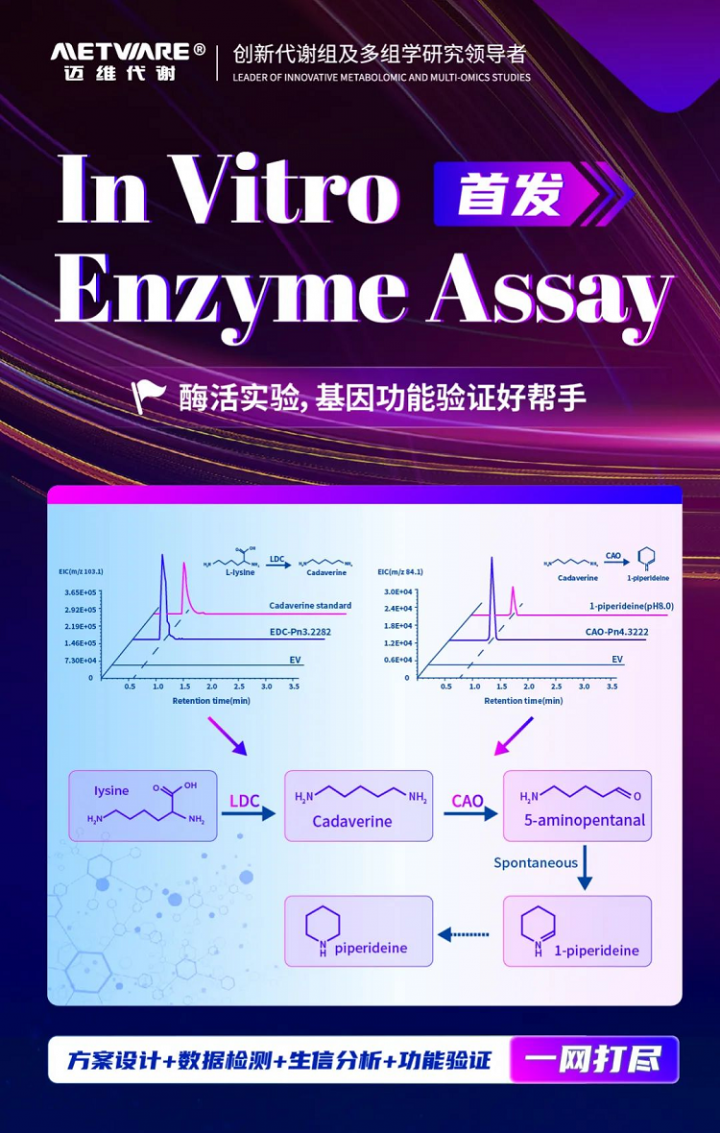
最近有两篇文章在朋友圈传播热度较高,一篇是海南大学罗杰教授2023年9月份发表在the plant journal上的metabolome profiling and transcriptome analysis filling the early crucial missing steps of piperine biosynthesis in piper nigrum l文章,文章通过转录组 代谢组对开花后2、4和6个月的黑胡椒的茎、叶、花和果实进行检测分析,鉴定了两个产生c5侧链支架阿魏酰二酮辅酶a的中间体的阿魏酰二酮-辅酶a合成酶(fds1和fds2),这是从苯丙素途径分支到胡椒碱生物合成的第一步和重要步骤。通过in vitro enzyme assay实验验证了由赖氨酸衍生的胡椒碱生物合成的前两个关键酶,赖氨酸脱羧酶(ldc)和铜胺氧化酶(cao),这些酶催化产生尸胺和1-哌啶。
第二篇文章是上海辰山植物园徐萍课题组2023年11月份发表在molecular plant上的multi-omics analyses of two leonurus species illuminate leonurine biosynthesis and its evolution文章,基因组学、转录组学和代谢组学的关联分析揭示了益母草碱合成路径,并确定了关键的候选基因,筛选和通过in vitro enzyme assay
实验鉴定了合成途径中的几个关键酶,包括精氨酸脱羧酶(adc)、udp-葡萄糖基转移酶(ugt)以及丝氨酸羧肽酶样(scpl)酰基转移酶。
in vitro enzyme assay实验在高分文章中频频出现,无论是基于现有技术手段的植物分子机制的研究,还是植物合成生物学目前越演越烈,in vitro enzyme assay实验无疑都是研究中最重要的分子实验手段,那么in vitro enzyme assay实验到底是什么,他能解决我们研究中的什么问题,我们下面细细展开。
1.in vitro enzyme assay--基因体外的完整功能实现
遗传信息的传递遵循着中心法则,由dna转录成rna,rna翻译成蛋白,蛋白作为酶去催化代谢物质的合成和降解。in vitro enzyme assay实验实现了体外结构基因的克隆,蛋白表达,和代谢物的催化过程。其中蛋白表达的过程是在原核体系/真核体系中诱导催化,通过质谱去检测孵化产物。
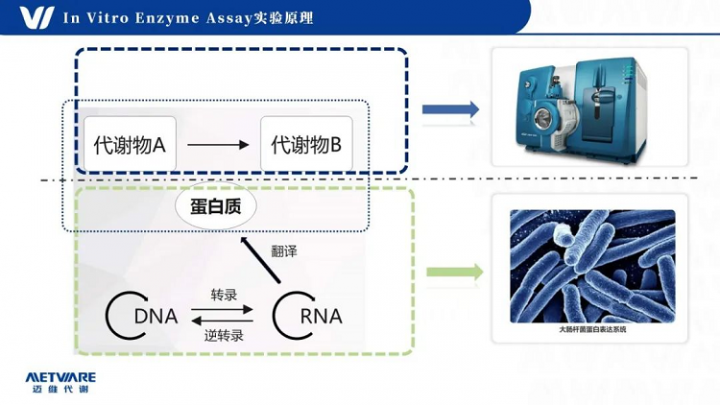
2.in vitro enzyme assay定义
通过将目的基因转入载体,后导入工程菌,诱导其表达获得有功能活性的蛋白,并构建含有该蛋白和底物的反应体系进行反应,以验证蛋白是否具有目标代谢物的催化作用。
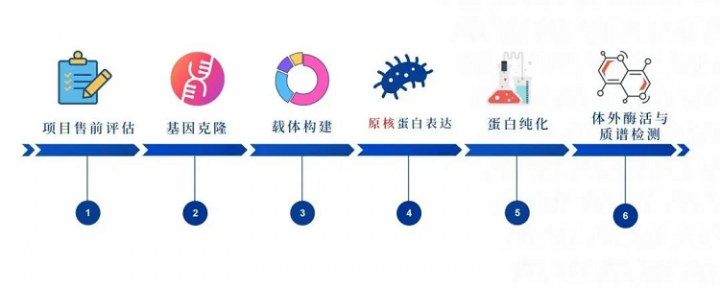
3.in vitro enzyme assay项目流程
in vitro enzyme assay项目流程包括项目售前评估,基因克隆,载体构建,原核蛋白表达,蛋白纯化,与体外酶活与质谱检测部分。
4.in vitro enzyme assay适用研究范围
in vitro enzyme assay适用于常规多组学研究之后,通过数据分析找到的候选基因进行后续的实验验证。组学的数据分析,只能是推测,要想确证筛选到的候选基因是否具有预测的功能只能通过分子实验手段去验证,可以通过遗传转化体系去验证,或者通过in vitro enzyme assay实验去单独验证某个基因的催化功能,但是大部分物种是缺乏稳定转化的遗传体系的,因此in vitro enzyme assay实验是一个周期短,不筛选物种,亲民基因验证思路。
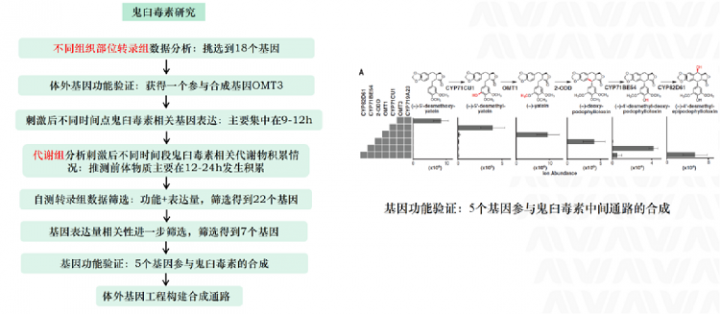
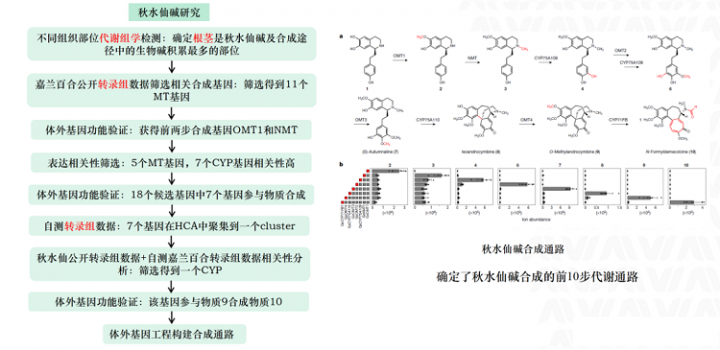
in vitro enzyme assay另一个适用方向是应用到中药活性物质合成通路探索,或者植物合成生物学的研究中。2015年发表在science上的鬼臼毒素代谢通路的研究文献,以及2020年发表在nature上的秋水仙碱的代谢通路的研究文献,研究思路均是通过转录组的技术手段筛选通路的候选基因,最后通过in vitro enzyme assay实验分步去验证基因的功能,从而打通整个中药活性物质的代谢通路,并进行体外基因工程构建,实现工程菌批量生产现有产量较低的代谢产物。
5.in vitro enzyme assay产品重磅上线
迈维代谢自主研发in vitro enzyme assay产品,打通了蛋白纯化,底物孵化至质谱检测的桥梁。产品重磅上市,意向客户可对接驻地技术或扫码沟通。
文献参考
1.lv y, zhu j, huang s, et al. metabolome profiling and transcriptome analysis filling the early crucial missing steps of piperine biosynthesis in piper nigrum l[j]. the plant journal, 2023.
2.li p, yan m x, liu p, et al. multi-omics analyses of two leonurus species illuminate leonurine biosynthesis and its evolution[j]. molecular plant, 2023.
3.reed j, orme a, el-demerdash a, et al. elucidation of the pathway for biosynthesis of saponin adjuvants from the soapbark tree[j]. science, 2023, 379(6638): 1252-1264.
4.lau w, sattely e s. six enzymes from mayapple that complete the biosynthetic pathway to the etoposide aglycone[j]. science, 2015, 349(6253): 1224-1228.
5.nett r s, lau w, sattely e s. discovery and engineering of colchicine alkaloid biosynthesis[j]. nature, 2020, 584(7819): 148-153.